Universidad de Puerto Rico
Recinto de Rio Piedras
Semestre: primer semestre 2018-2019
Codificación: MATE 4995 Sección 012
Título del curso- Análisis de datos masivos en aplicaciones biomédicas I (BBD1)
Profesora- Dra. Maria E. Perez
Requisitos- MATE 3026 o equivalente y CCOM 3030 o permiso de la profesora.
Este curso busca preparar al estudiante para dos objetivos fundamentales:
- Poder trabajar con grandes cantidades de datos. Algo esencial en futuros aspectos de todo tipo de investigación.
- Poder participar en el proyecto de IDI-BD2K el próximo verano con experiencias de investigación en los Centro de Excelencia de BD2K en las universidades de Harvard, Pittsburgh y la Univ. de California, Santa Cruz.
Estudiantes de cualquier bachillerato de Ciencias Naturales pueden matricularse.
Mas información sobre el proyecto IDI-BD2K
Hoy como nunca antes la investigación biomédica está generando cantidades masivas de datos, cuyo análisis e interpretación tiene el potencial de producir dramáticos avances en nuestro conocimiento sobre la salud humana y sobre nuestra calidad de vida. El análisis de estos conjuntos masivos de datos (“Big Data”) requiere técnicas que combinan conocimientos en Biología, Química, Estadística, Ciencias de Cómputo y otras áreas.
El proyecto IDI-BD2K estará ofreciendo el curso MATE 4995 Sección 23 – Análisis de datos masivos en aplicaciones biomédicas I (BBD1) en otoño 2017. En este curso podrás aprender cómo encontrar grupos de genes sobreexpresados en una condición, como el cáncer. Puedes aprender a crear e interpretar modelos lineales que describen la respuesta a un tratamiento. Verán cómo manejar conjuntos masivos de datos genómicos y analizarlos.
Estudiantes que completen este curso y su continuación BBD2 calificarán para ir a un internado en alguno de los Centros de Excelencia de BD2K como Harvard, University of California Santa Cruz, y Pittsburgh.
Para información adicional puedes comunicarte con la Dra. Perez: maria.perez34@upr.edu
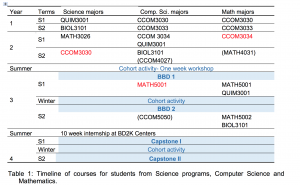
Si aun no estas preparado para tomar BBD1 y 2, asegurate que estas tomando los cursos sugeridos por los Centros de Excelencia BD2K para tu concentración consultando la tabla a continuación: